Exploration des génomes de soya
0
0
0
Analyse de la variation nucléotidique et structurale chez le soya par une approche de re-séquençage
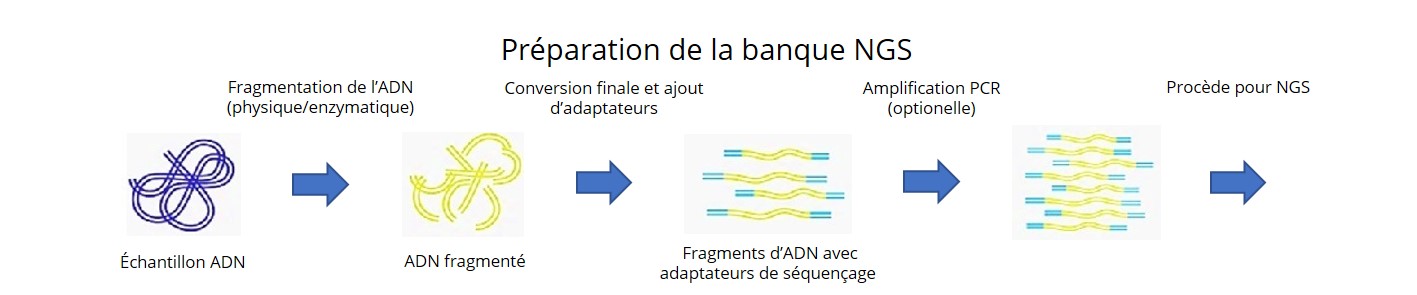
Cette étude présente un séquençage d’un ensemble de 102 lignées de soya hâtif, représentatif du soya canadien. Nous avons obtenu une couverture exhaustive de la diversité nucléotidique et de la variation structurelle (SV) chez le soya.
- Nous avons détecte près de 5M variants nucléotidique (SNP, MNP et indels) avec un haut niveau d’exactitude (98,6%).
- Nous avons utilisé ce catalogue de SNP comme panel de référence pour imputer des génotypes manquants (un-typed genotypes) dans des ensembles de données issus d'outils de génotypage de faible densité (150K GBS-SNP / 530 échantillons), avec un haut niveau d’exactitude (96,4%).
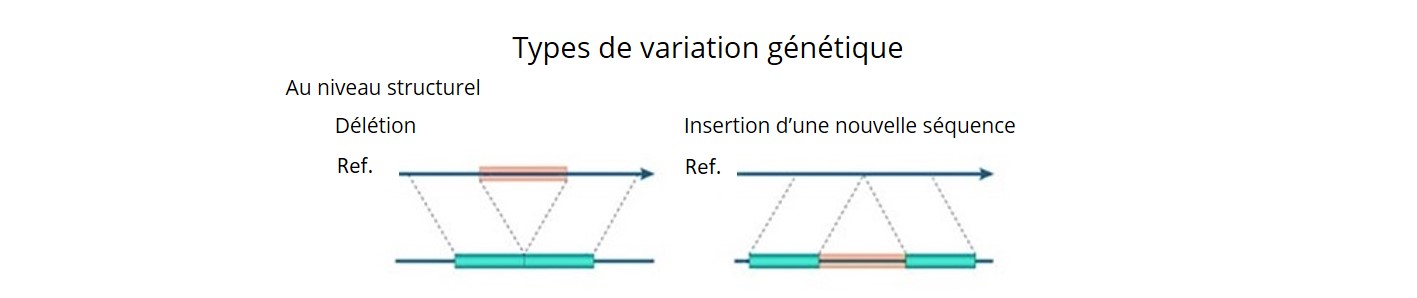
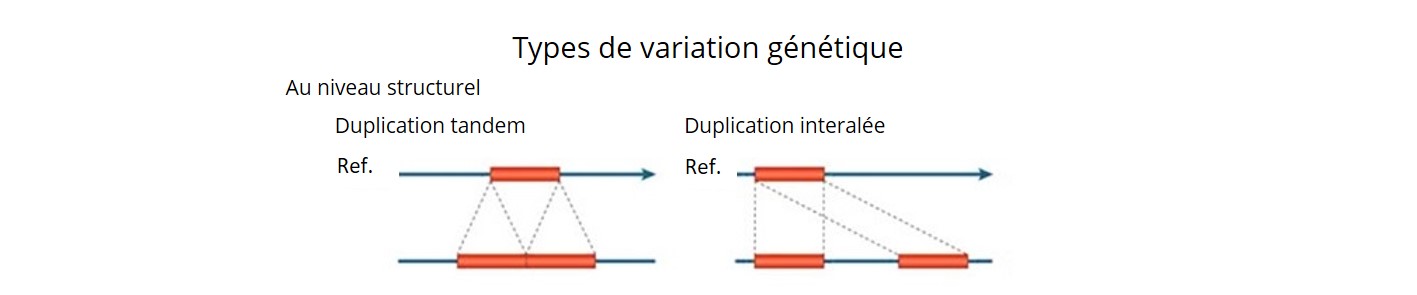
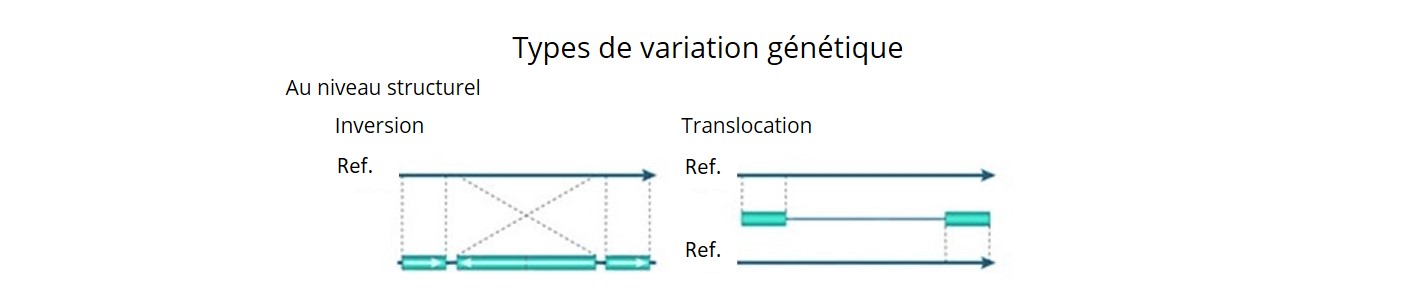
- En utilisant une combinaison de trois pipelines bioinformatiques, nous avons découvert ~ 92 K SV (délétions, insertions, inversions, duplications, CNV (copy number variation ou variation du nombre de copies en français) et translocations) avec un niveau de précision de 90%.
Cette étude présente la première description complète de la diversité nucléotidique et haplotypique, en plus des variants structuraux, qui a été réalisée chez un sous-ensemble d'une grande culture.